Куратор курса: к.б.н. Игнатьева Елена Васильевна
| Бакалавриат | Магистратура | |
|---|---|---|
| Аннотация рабочей программы дисциплины | Скачать | Скачать |
| Рабочая программа | Скачать | Скачать |
| Фонд оценочных средств | Скачать | Скачать |
| Трудоемкость дисциплины | 2 З.Е. (72 ч.) | |
| Форма промежуточной аттестации | зачет | |
Содержание дисциплины включает изложение современных представлений о молекулярных механизмах регуляции экспрессии генов, роли регуляторных геномных последовательностей в их реализации, а также о базах данных и компьютерных методах анализа данных из этой области знаний.
Перечень основных разделов
1. Регуляция транскрипции и регуляторные геномные последовательности
2. Регуляция трансляции и регуляторные геномные последовательности
3. Компьютерный анализ регуляторных геномных последовательностей: методы и алгоритмы
Тематический план курса
- Механизмы регуляции транскрипции (к.б.н. Игнатьева Е.В.) – 3 лекции
- Базы данных по регуляции транскрипции (к.б.н. Игнатьева Е.В.) – 2 лекции
- Конформационные и физико-химические контекстно зависимые свойства ДНК и их использование для построения методов анализа и распознавания функциональных районов ДНК. Метод конформационных параметров: система B-DNA Video (к.б.н. Ощепков Д.Ю.) — 1 лекция
- Регуляция экспрессии генов на уровне трансляции (к.б.н. Игнатьева Е.В.) — 2 лекции
- Распознавание и анализ сайтов связывания транскрипционных факторов (к.б.н. Вишневский О.В.) — 1 лекция
- База данных по регуляции транскрипции TRRD (к.б.н. Игнатьева Е.В.) – 1 практическое занятие
- Компьютерные ресурсы, позволяющие анализировать регуляторные последовательности мРНК (к.б.н. Игнатьева Е.В.) – 1 практическое занятие
Вопросы для подготовки к зачету
1. Транскрипция – ключевой этап экспрессии гена, функции РНК-полимеразы, основные этапы транскрипции.
2. Транскрипция у прокариот, характеристика основных регуляторных белков и регуляторных последовательностей.
3. Известны ли отличия по структуре для промоторных районов генов E.coli, инициация транскрипции которых происходит при участии разных сигма факторов? Какие это отличия?
4. Особенности транскрипции у эукариот. РНК-полимеразы и гены, транскрибируемые каждой полимеразой.
5. Субъединичный состав РНК-полимераз эукариот. Доменная организация РНК-полимеразы II и роль доменов в реализации функции РНК-полимеразы.
6. Основные этапы транскрипционного цикла, осуществляемого РНК-полимеразой II. Роль С-концевого домена РНК-полимеразы II в транскрипционном цикле.
7. Основные классы белков, регулирующих транскрипцию эукариот, и их краткая характеристика.
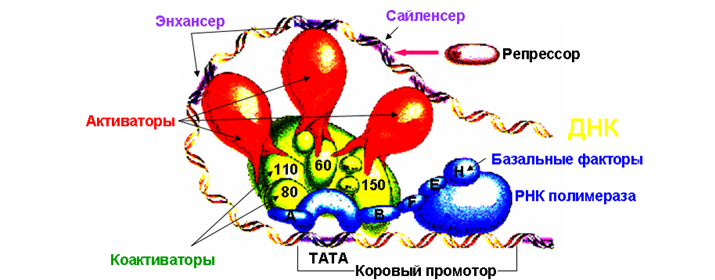
8. Базальные транскрипционные факторы (=General transcription factors, GTFs). Базальный транскрипционный комплекс, модели его формирования.
9. Чем отличаются транскрипционные факторы (сайт-специфичные) от базальных транскрипционных факторов? В чем разница между корегуляторными белками и белками медиаторного комплекса?
10. Регуляторные элементы и регуляторные единицы (районы), контролирующие транскрипцию генов эукариот.
11. Что такое коровый промотор и какова его локализация? Чем отличается коровый промотор от промоторного района ?
12. Структура промотора эукариот и его основные элементы.
13. Транскрипционные факторы, два варианта их классификации, пути активации.
14. Остановка (пауза) РНК-полимеразы II на стадии элонгации — дополнительный этап регуляции транскрипции
15. Влияние нуклеосомной укладки ДНК на интенсивность транскрипции. Механизм регуляции плотности нуклеосомной укладки в зависимости от высокого/низкого уровня ацетилирования гистонов.
16. Модификации хроматина, влияющие на интенсивность транскрипции генов эукариот. В чем их сущность?
17. Код модификаций хроматина – что это такое? Сложность кода модификаций хроматина.
18. Локус-контролирующие районы, MAR и инсуляторы, их влияние на транскрипцию.
19. Роль транскрипционных факторов CTCF и когезинового белкового комплекса.
20. Что такое энхансерная РНК? Регуляторные функции энхансерной РНК.
21. Три модели взаимодействия между регуляторными участками генов (промоторами и энхансерами) в геноме человека (по данным анализа эксперимента с применением методики ChIA-PET). Какие регуляторные элементы на ДНК и какие регуляторные белки определяют трехмерную структуру хроматина?
22. Хромосомный оперон (chro-operon or chroperon). Как эта структура влияет на интенсивность транскрипции?
23. На примере механизма активации гена интерферона бета человека перечислить классы регуляторных белков, участвующих в подготовке корового промотора к взаимодействию с прединициаторным комплексом (ПИК).
24. Базы данных по регуляции транскрипции. Какие сущности могут быть охарактеризованы в базах по этой тематике? Охарактеризовать одну из известных баз (на выбор, кроме TRRD).
25. Базы данных по регуляции транскрипции. «Критерии полезности баз данных» Охарактеризовать одну из известных баз (на выбор, кроме TRRD).
26. Базы данных по регуляции транскрипции. Охарактеризовать одну из известных Вам баз, содержащую информацию по регуляторным последовательностям на ДНК (кроме TRRD).
27. Базы данных по регуляции транскрипции. Охарактеризовать одну из известных Вам баз по белкам, регулирующим транскрипцию.
28. Базы данных по регуляции транскрипции. Что содержится в базах данных по матрицам сайтов связывания транскрипционных факторов. Для чего нужны такие базы?
29. Характеристика базы TRRD. Типы данных, аккумулированных в TRRD, возможности поиска данных.
30. Чем определяется различие конформационных параметров между динуклеотидами?
31. Каким образом принято описывать взаимное расположение пар нуклеотидов относительно друг друга? Связь между системой координат и основными конформационными параметрами.
32. Классификация ДНК-связывающих доменов транскрипционных факторов. Опишите суперклассы ДНК-связывающих доменов.
33. Правила Калладина.
34. Оценка качества работы программ предсказания сайтов связывания.
35. Метод реализаций.
36. Метод Gibbs sampler.
37. Метод локального множественного выравнивания регуляторных последовательностей на примере программы CONSENSUS. Не путать с методом консенсуса для описания уже выровненной выборки сайтов связывания!
38. Метод к-плетов.
39. Метод консенсуса и матричные методы описания и распознавания сайтов связывания транскрипционых факторов.
40. Перечислить особенности трансляции у прокариот. Сколько факторов инициации трансляции Вам известно у прокариот?? — у эукариот??
41. Перечислить особенности трансляции у эукариот. Сколько факторов инициации трансляции Вам известно у прокариот?? — у эукариот??
42. Два механизма инициации трансляции у эукариот.
43. Что такое IRES (internal ribosome entry site)? Особенности функционирования IRES.
44. Каков биологический смысл существования альтернативного способа инициации трансляции с участием IRES?
45. Контекстные характеристики лидерной последовательности мРНК (=5’-НТП=5’-НТО=5’-UTR), влияющие на эффективность трансляции у эукариот. Что позволяет предсказывать программа AUG_hairpin ?
46. Сигналы экспрессии, которые обеспечивают пост-транскрипционный контроль экспрессии. Что это такое? Перечислить известные Вам виды сигналов. Описать функционирование одного из сигналов (любого, на выбор).
47. Описать функционирование сигналов экспрессии, регулирующих стабильность мРНК, на примере IREs (iron-responsive elements).
48. Сигналы экспрессии, обеспечивающие транспорт мРНК к месту трансляции. Привести два примера, когда необходима регуляция транспорта мРНК.
49. Что такое микроРНК, и каким образом осуществляется функция микроРНК?
50. Где находится последовательности «Seed» и MRE? Каковы их функции?
51. Рибопреключатель (riboswitch). Описать функционирование на примере тиаминпирофосфатного переключателя. Какие функциональные домены входят в состав переключателя?
52. С какими базами данных и программами, относящимися к теме «Трансляция», Вы познакомились? Расскажите про одну из баз или программ (на выбор).
Литература
- Альбертс Б., Брэй Д., Льюис Дж., Рэфф М., Робертс К., Уотсон Дж. Молекулярная биология клетки. М: Мир, 1994. Т. 1, разд. 3.2 «Нуклеиновые кислоты»; т. 2, разд. 10.1 «Стратегия генетического контроля», 10.2 «Контроль инициации транскрипции».
- Льюин Б. Гены. М.: БИНОМ. Лаборатория знаний, 2011.
- Меркулова Т. И., Ананько Е. А., Игнатьева Е. В., Колчанов Н.А. Регуляторные коды транскрипции геномов эукариот // Генетика. 2013. Т. 49, № 1. С. 37–54.
- Сингер М., Берг П. Гены и геномы М: Мир, 1998. Т. 1, разд. 3.1–3.2. «Основные положения процесса экспрессии генов», «Транскрипция: передача информации о нуклеотидной последовательности ДНК на уровень РНК».
Дополнительная литература
- Вингендер Э. Классификация транскрипционных факторов эукариот // Молекулярная биология. 1997. Т. 31 (4). С. 584–601.
- Колчанов Н. А., Подколодная О. А., Ананько Е. А., Игнатьева Е. В., Подколодный Н. Л., Меркулов В. М., Степаненко И. Л., Поздняков М. А., Белова О. Е., Григорович Д. А., Наумочкин А. Н. Регуляция транскрипции генов эукариот: описание в базе данных TRRD // Молекулярная Биология. 2001. Т. 35 (6). С. 934–942.
- Bajic V. B., Chong A., Seah S. H., Brusic V. Intelligent system for vertebrate promoter recognition // IEEE Intelligent Systems. 2002. V. 17 (4), p. 64-70.
Дополнительные материалы
- Intelligent system for vertebrate promoter recognition 2002 (скачать)
- The biology of eukaryotic promoter prediction — a review_1999 (скачать)
- Регуляторные коды транскрипции геномов эукариот (2013) (скачать)
- Регуляторные элементы эукариотического генома, контролирующие транскрипцию (2015) (скачать)
- Компартментализация клеточного ядра и пространственная организация генома (2015) (скачать)
- Регуляторная геномика – экспериментально-компьютерные подходы (2015) (скачать)
Лекции 2022
| Название | Ссылка на видео | Презентации |
|---|---|---|
| Лекция №1. Механизмы регуляции транскрипции (лектор к.б.н. Игнатьева Е.В.) | Доступ ограничен | Доступ ограничен |
| Лекция №2. Механизмы регуляции транскрипции ч. 2 (лектор к.б.н. Игнатьева Е.В.) | Доступ ограничен | Доступ ограничен |
| Лекция №3. Механизмы регуляции транскрипции ч. 3 (лектор к.б.н. Игнатьева Е.В.) | Доступ ограничен | Доступ ограничен |
| Лекция №4. Механизмы регуляции транскрипции ч. 4 (лектор к.б.н. Игнатьева Е.В.) | Доступ ограничен | Доступ ограничен |
| Лекция №5. Распознавание и анализ сайтов связывания транскрипционных факторов (лектор к.б.н. Вишневский О.В.) | Доступ ограничен | Доступ ограничен |
| Лекция №6. Конформационные и физико-химические контекстно зависимые свойства ДНК и их использование для построения методов анализа и распознавания функциональных районов ДНК. Метод конформационных параметров: система B-DNA Video и SITECON (лектор к.б.н. Ощепков Д.Ю.) | Доступ ограничен | Доступ ограничен |
| Лекция №7. Регуляция экспрессии генов на уровне трансляции ч.1 (лектор к.б.н. Игнатьева Е.В.) | Доступ ограничен | |
| Лекция №8. Регуляция экспрессии генов на уровне трансляции ч.2(лектор к.б.н. Игнатьева Е.В.) | Доступ ограничен | Доступ ограничен |
| Лекция №9. Транскрипция. Базы данных по регуляции транскрипции (лектор к.б.н. Игнатьева Е.В.) | Доступ ограничен | Доступ ограничен |
| Лекция №10. Базы данных по регуляции транскрипции (лектор к.б.н. Игнатьева Е.В.) | Доступ ограничен | Доступ ограничен |